Cronograma
Los resúmenes se aceptarán desde el 1 marzo al 3 de abril.
Inscripciones abiertas hasta el 15 de abril.
Auspiciantes


Talleres
¿Qué es?
La III Conferencia Amazónica de Herencia, Biodiversidad y Ambiente, que se realizará en la ciudad de Tena-Ecuador, es un espacio para compartir ideas y experiencias, que sé efectúa cada año con la finalidad de reunir a participantes de todo el mundo en la Amazonía ecuatoriana para profundizar en los cambios que ha sufrido el ambiente durante los últimos años, su repercusión en la pérdida de biodiversidad y potenciales efectos en la herencia poblacional.
La conferencia es organizada por docentes y estudiantes de la Universidad Regional Amazónica Ikiam, está dirigida al público en general, estudiantes, docentes, investigadores, empresarios, cualquier persona interesada en aprender o actualizar conocimientos asociados a herencia, biodiversidad y ambiente.
Este evento se ejecuta de manera presencial durante 4 días, donde se llevarán a cabo conferencias magistrales a cargo de científicos ecuatorianos y extranjeros con amplios conocimientos sobre herencia genética, epigenética, biodiversidad, variabilidad genética, ambiente, salud humana, entre otros. Te invitamos a ser parte de esta conferencia que se efectuará del 24- 27 de abril 2023 en Tena-Ecuador, puedes inscribirte como asistente o expositor.
Objetivos
Contribuir con la formación académica y científica de los participantes al conocer los avances científicos y tecnológicos en el área de la ingeniería genética y de poblaciones, enmarcadas en la biodiversidad y conservación ambiental.
Crear redes de colaboración académica y científica en el ámbito de la biotecnología, epigenética, protección ambiental, para proponer respuestas a los problemas actuales.
Promover la transformación en el desarrollo sostenible, formando profesionales ambiental y socialmente responsables, con sólidos conocimientos científicos y tecnológicos.
Apoyar al conocimiento y mantenimiento de la biodiversidad, así como el respeto al territorio que nos rodea.
Temas
Tecnologías
Técnicas
Convocatoria de revista abierta
Los mejores trabajos presentados en el evento pueden ser parte de la siguiente edición de la revista Científica y Tecnología UPSE, una vez que cumplan con los requisitos de la misma.

His research leverages genomic technologies to identify functionally important regions of the genome. Recent research has focused on the evolution of wing patterns in Heliconius butterflies. The enormous wing pattern variation in the group offers exceptional opportunities for genomic level studies designed to reveal how morphological variation is created through development and modified by natural selection within the context of an extraordinary adaptive radiation. McMillan maintains an active molecular genetic lab, as well as experimental facilities for Heliconius culture and study.

Universidad de Hiroshima, Japón PhD Veterinary Medicine at University of Tokyo He is currently working for elucidating the species-dependent mechanisms of non-coding RNA-directed epigenome formation in mammals.

(Nottingham University, UK) Sian Bray is fascinated by evolution, with skills including ‘dry-lab’ bioinformatic and programming skills as well as ‘wet-lab’ biochemistry and structural biology skills. She is involved in the design and delivery of Nottingham’s new MSc course in Bioinformatics.The main research interest of my lab is genomic and proteomic evolution in response to stressful or extreme conditions, via high-resolution large-scale genomic analyses, as well as protein structural modelling and functional predictions. This work has recently branched out into computational methods development via the work of my previous postdoc and a fruitful collaboration.

(Nottingham University, UK). His goal is to contribute substantially to our understanding of fundamental principles of evolutionary change. In particular, he is working to understand precisely the way that diverse population genomic processes lead to solutions to serious environmental challenges. His research group does this by applying large-scale population genomics to wild populations that have overcome clearly definable, quantifiable hazards. That recent advances allow us to unambiguously identify candidate natural alleles underlying particular adaptations is revolutionary, making this a great time to apply population genomics to wild species.

Ecole Pratique des Hautes Etudes, Francia Andras Paldi is biochemist and geneticist. He is professor at the École Pratique des Hautes Études in Paris. His research focuses on epigenetics and cell differentiation. He is working on the biological role of stochastic fluctuations of gene expression and the involvement of epigenetic mechanisms and the energetic metabolism in the process of hematopoietic cell differentiation.

Duke University, USA. I study the evolution of genes and genomes with the broad aim of understanding the origins of biological diversity. My approach focuses on changes in the expression of genes using both empirical and computational approaches and spans scales of biological organization from single nucleotides through gene networks to entire genomes. My research leverages the advantages of several different model systems, but primarily focuses on sea urchins and primates (including humans).

Universidad de las Américas, Chile. Coordinador de Instituto de Ciencias Naturales de Sede Concepción expone en Congreso Latinoamericano de Genética

Universidad de Washington, USA Evolutionary and Developmental genetics of butterflies and Moths

Universidad Austral, Chile My research focuses on questions related to the evolution of genomes, using vertebrate animals as a major study system, with special emphasis on understanding the dynamics of gene gain and losses and its connection with the origin of biological novelties.
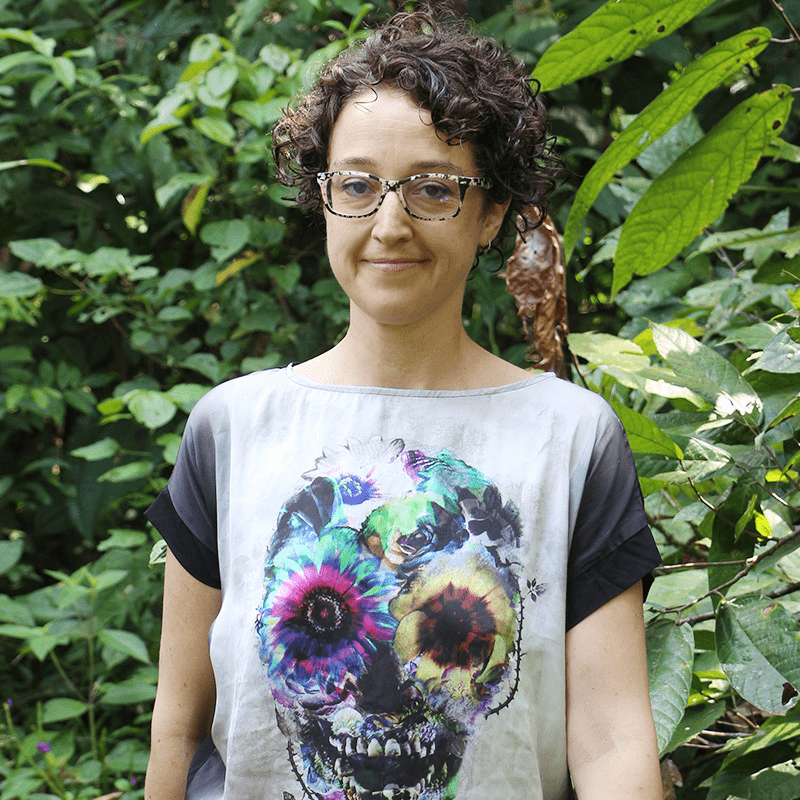
Universidad Regional Amazónica Ikiam, Ecuador Caroline Nicole Bacquet currently works at the Life Sciences Department, Universidad Regional Amazónica IKIAM. Caroline does research in Cell Biology, Molecular Biology and Genetics. Their current project is “Unraveling the mechanisms of brain and behavioral elaboration in ecologically diverse butterflies”.

(physics, mathematics) University of Nottingham Cyril Rauch obtained his BSc, MSc and DEA from the University of Jussieu (Paris – France) followed by his PhD from the Curie Instituteand University of Jussieu (Paris – France) devoting his time on the biophysics of the cell membrane . He then spent 3 years at the Royal Veterinary College (London – UK) working on mechanotransduction and one year at the School of Pharmacy and Pharmaceutical Science of the University of Manchester (UK) working on a new detection method regarding drug/cell interactions. Finally he joined the School of Veterinary Medicine and Science (University of Nottingham) in September 2006 to set up a group focusing on the Physical and Mathematical sides of Veterinary Medicine and Science.

Duke University, USA. My academic and professional expertise is in comparative genomics, bioinformatics, cognitive ecology, human biology, population genomics and evolutionary biology, with hands-on practice using classic molecular biology methods, Next Generation Sequencing (NGS) technologies and bioinformatics tools, with demonstrated scientific publication record. Now, I am using computational and analytical tools to discover the most extraordinary regions of the human regulome, I am fascinated with mapping and documenting the DNA sequences that makes us human.
Convocatoria de resúmenes
Convocatoria de inscripciones
Lugar y fecha del evento
Día del evento

PhD. en Biología Celular y Molecular de la Universidad Austral de Chile, es docente investigadora de la Universidad Regional Amazónica Ikiam en Ecuador.
Ha realizado investigaciones sobre expresión y regulación de genes en mamíferos.

Máster en Terapias Avanzadas e Innovación Biotecnológica de la Universidad Francisco de Vitoria, docente investigador de la Universidad Regional Amazónica Ikiam en Ecuador.
Ha hecho investigaciones sobre aislamiento y caracterización de células madre de mamíferos.

Estudiante de Biotecnología de la Universidad Regional Amazónica Ikiam en Ecuador, Ayudante de cátedra de Genética y Biología Molecular II.
Centró su tesis en el estudio del efecto del estrés térmico sobre la expresión de genes de proteínas de shock térmico en mariposas tropicales.
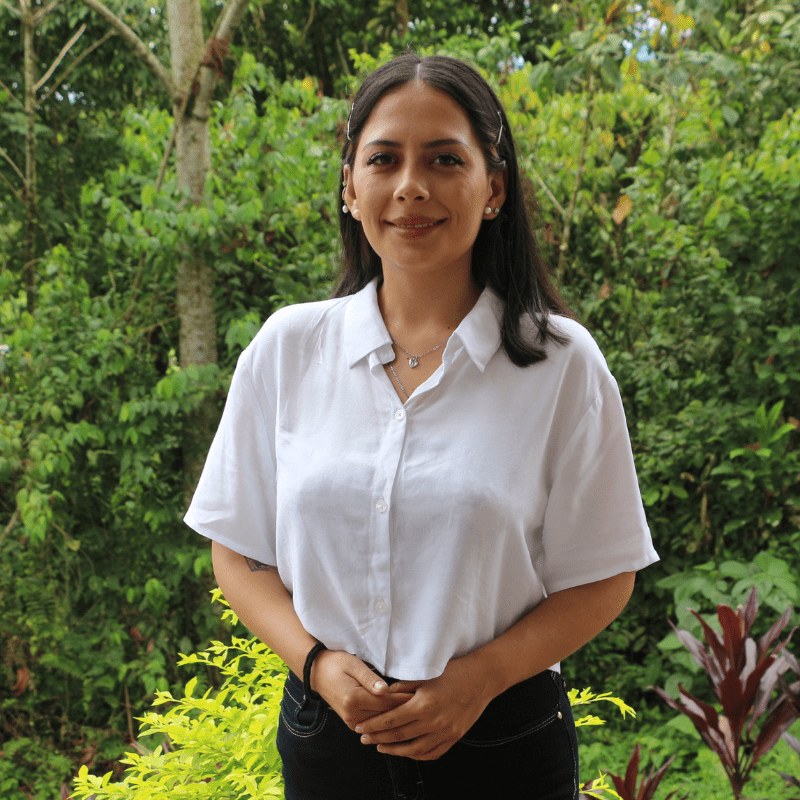
Estudiante de Biotecnología de la Universidad Regional Amazónica Ikiam en Ecuador, ayudante de cátedra de Biología Molecular I.
Se encuentra efectuando su tesis sobre el aislamiento de péptidos con actividad antimicrobiana en la secreción de la piel de anfibios.

Estudiante de Biotecnología de la Universidad Regional Amazónica Ikiam.

Ing. en Biotecnología Ambiental, MSc. en Ingeniería Ambiental, egresado de la Maestría en Cambio Climático, Sustentabilidad y Desarrollo. Docente en la Universidad Regional Amazónica IKIAM; miembro del Biomass to Resourse group y la Red de Análisis de Ciclo de vida de Ecuador.

Ingeniero en Biotecnología graduado en Ikiam. Fue ayudante de Cátedra de Biología Molecular II y Genética. Miembro de la SMBE.
En su tesis de pregrado estudió los efectos del estrés térmico en la expresión génica de Hsp70 en mariposas.

Estudiante de Biotecnología de la Universidad Regional Amazónica Ikiam.
Está realizando su tesis de pregrado centrado en la metilación del genoma de mariposas tropicales.
Dirección: parroquia Muyuna, kilómetro 7 vía a Alto Tena
Teléfono: 06 370 0040
Email: secretaria.general@ikiam.edu.ec
© Ikiam Copyright 2022. All Rights Reserved. – Powered by DTI